Notre expertise s’étend jusqu’à l’analyse bioinformatique et/ou bio-statistique de vos données. Nous avons mis en place différents pipelines d’analyse automatisés, basés sur les meilleurs algorithmes du moment. Nous disposons de plusieurs dizaines de serveurs de calcul pour les faire tourner, ainsi que des capacités de stockage nécessaires.
Analyse bioinformatique et biostatistique
Les volumes et la qualité des données produites par les nouvelles générations de séquenceurs changent régulièrement. Notre laboratoire s’adapte continuellement et propose le développement d’utilitaires pour vous répondre et obtenir le meilleur de vos données : alignement et découverte de variants par le séquençages d’exomes et de génomes, annotation des variants découverts à partir de données publiques (Ensembl, Encode, …), études familiales et épigénétiques telles que l’étude de transcriptomes ou de méthylomes et d’interactions protéines-ADN. Nous produisons également diverses statistiques vous assurant le contrôle de la qualité des données générées.
Le groupe de biostatistique est composé de 3 biostatisticiens. Il assure :
Expertise
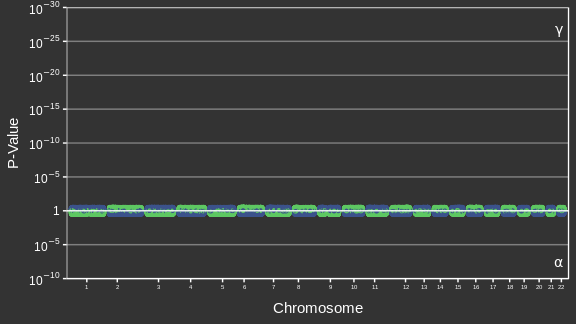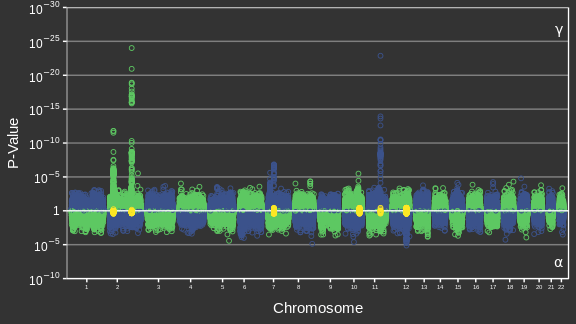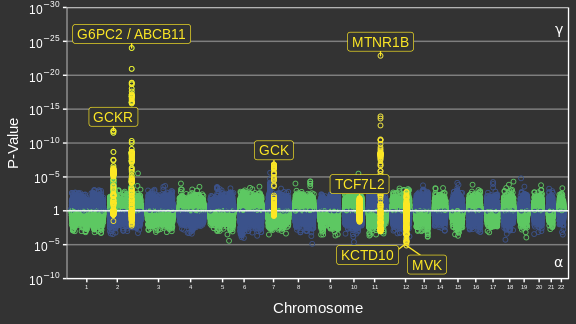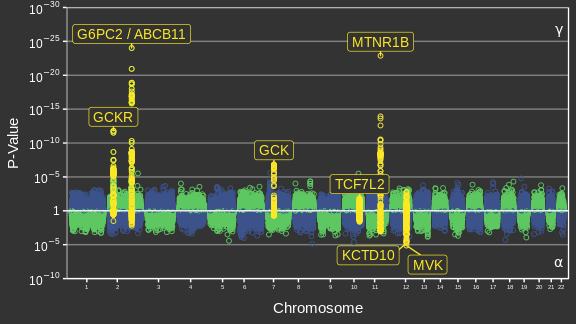
Adaptée de Canouil et al. (2018)
Pipelines & Extension R (https://github.com/umr1283/)
“Detection of Clonal Mosaic Events” : http://www.good.cnrs.fr/app/uploads/2012/09/CALLME.zip
snpEnrichment : https://CRAN.R-project.org/package=snpEnrichment
clere : https://CRAN.R-project.org/package=clere
NACHO : https://m.canouil.fr/NACHO/, https://CRAN.R-project.org/package=NACHO
insane : https://m.canouil.fr/insane/, https://CRAN.R-project.org/package=insane
Le groupe de bioinformatique est composé de 4 bioinformaticiens. Il assure :
Nos principaux projets de développements futurs sont :

